Wetenschappers van onderzoeksbedrijf KeyGene in Wageningen zijn als eerste naar buiten gekomen met een rapport waarin zij vertellen dat ze erin geslaagd zijn om zogenaamde (= selectieve) DNA-sequencing met behulp van de Read Until-tool van Oxford Nanopore Technologies uit te voeren. De techniek maakt vaker sequencen van belangrijke DNA-regio's mogelijk en geeft daarmee meer kennis.
Het team combineerde een Oxford Nanopore Technologies MinION DNA-sequencer, een krachtige met GPU-uitgeruste computer en de Read Until-software van Oxford Nanopore voor het analyseren van meloen-DNA. Op die manier was het mogelijk om 800 vooraf gekozen belangrijke regio’s in het meloen-DNA, maar liefst acht keer zo vaak te sequencen dan de controle, waarbij de Read Until software niet werd gebruikt. Doordat deze regio’s vaker geanalyseerd werden, kon het DNA met een veel grotere zekerheid in kaart gebracht worden.
Met deze doorbraak, zo noemt KeyGene deze stap, kunnen plantenveredelaars beter veredelen op belangrijke eigenschappen, zoals smaak, vruchtkleur, houdbaarheid en ziekteresistentie. Door de regio’s in het planten-DNA die bij deze eigenschappen betrokken zijn beter in detail te sequencen en te analyseren, kunnen veredelaars variatie beter begrijpen en gebruiken voor selectie van nieuwe rassen. Op deze manier kunnen verbeterde rassen sneller op de markt worden gebracht.
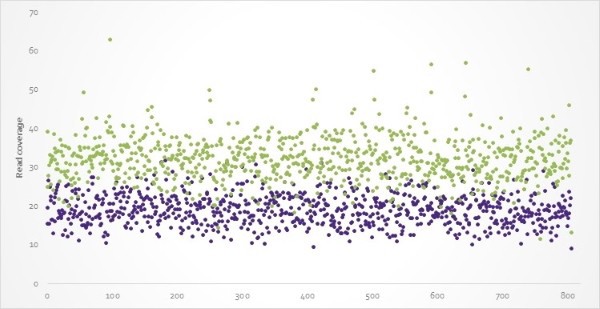
De 800 gewenste gebieden van meloen-DNA werden bij gebruik van de Read-Until tool (groene stippen) veel vaker gesequenced dan met niet-selectieve sequencing (paarse stippen)
Technologie
Met adaptieve sequentiebepaling kan de selectie van DNA-regio's worden gesequenced. Wanneer een DNA-fragment door een porie van een Oxford Nanopore-sequencer loopt, genereert het apparaat een reeks DNA-bouwstenen. Een krachtige computer vergelijkt die reeks ‘realtime’, dus op het moment dat het fragment door de porie gaat, met de informatie over de regio's waarin de onderzoeker of veredelaar in geïnteresseerd is.
Als de computer concludeert dat het fragment geen deel uitmaakt van een van één van de vooraf ingegeven gewenste regio’s van het DNA, krijgt de porie het signaal om het DNA-fragment terug te duwen naar het monster, vóórdat het volledige fragment wordt geanalyseerd. De porie is dan weer beschikbaar voor een volgend DNA-fragment uit het monster.
Dit gebeurt allemaal in slechts een fractie van een seconde. Het vereist een gelijktijdige real-time vergelijking tussen de sequentie van de DNA-fragmenten die door álle 512 poriën in de DNA sequencer lopen, met álle sequenties van geselecteerde interessegebieden in het DNA van de plant.
Menselijk DNA v.s. planten DNA
Recent onderzoek heeft aangetoond dat het inderdaad mogelijk is om op deze manier gericht bepaalde gebieden van menselijk DNA selectief te sequencen. Plant-DNA heeft zijn eigen uitdagingen, zoals de grote hoeveelheid repetitief DNA: DNA waarbij hetzelfde patroon van DNA-bouwstenen (nucleotiden), keer op keer herhaald wordt. Tevens zijn veel planten genomen vele malen groter dan het menselijke DNA waardoor deze techniek extra van pas komt.
Daarom vroegen KeyGene-wetenschappers zich af of ze het ook zou lukken met planten-DNA. Ze kozen ervoor om het genoom van meloen als testcase te gebruiken, omdat dit DNA al goed in kaart is gebracht. Ze ‘laadden’ de computer/database met de DNA-sequenties van 800 regio's van het meloen-DNA waarvan ze selectief de sequentie wilden bepalen. De computer moest dus continu alle 512 DNA-fragmenten in de poriën van de sequencing-machine vergelijken met de 800 gebieden van meloen-DNA die in het geheugen van de computer waren opgeslagen.
Acht keer beter
Het team was inderdaad in staat om de sequentiebepalingscapaciteit van de DNA-sequencer beter te gebruiken voor de regio’s waar ze in geïnteresseerd waren. Het lukte ze om die stukken acht keer zo vaak te laten sequencen, waardoor de bouwsteenvolgorde sneller en met grotere zekerheid kon worden bepaald.
Het succes maakt duidelijk dat selectief DNA sequencen het gemakkelijker en minder tijdrovend zal maken wanneer men bijvoorbeeld belangrijke DNA-regio's van verschillende rassen en verwanten van gewassen met elkaar wil vergelijken. Sequencing-expert Alexander Wittenberg: ‘‘Read Until is een fantastische applicatie die geen specifieke monstervoorbereiding vereist, nieuwe mogelijkheden opent in onderzoek naar gewas-innovaties en de kosten verder verlaagt. "
Om andere wetenschappers ook te laten profiteren van het werk en de bevindingen bij KeyGene, heeft het team een ‘White paper’ opgesteld, waarin de aanpak en de resultaten gedetailleerder worden beschreven.
Bij KeyGene zal de doorbraak worden gebruikt in het onderzoek voor de ontwikkeling van innovaties voor het agro food domein.
Voor meer informatie: KeyGene
KeyGene
www.keygene.com
